2024年4月10日,Nature子刊《Nature Computational Science》在线发表了重庆大学大数据与软件学院弘深青年教师曾远松关于单细胞ATAC数据细胞注释的论文。该论文第一作者为弘深青年教师曾远松博士,通讯作者为中山大学杨跃东教授。

单细胞ATAC测序技术(scATAC-seq)为揭示细胞类型的异质性开辟了新的途径。然而,细胞测序数据存在高维度和高稀疏性,导致scATAC-seq数据的细胞精确注释充满挑战。此前一些注释方法尝试通过计算基因内的ATAC峰值,并依据特定基因的峰值数量来识别细胞类型;但这些方法由于未能充分考虑基因内ATAC峰的特异性,导致其准确性受限。实际上,ATAC峰的调控功能依赖于其基因组序列是否含有可及的、细胞类型特有的增强子区域以及转录因子的结合位点,这些元素是推断细胞特性及其发育阶段的关键线索。因此,能否根据ATAC峰准确学习这些内在的DNA序列调控模式,并用于区分细胞类型?
SANGO是一个准确且可扩展的基于图神经网络的单细胞ATAC数据注释方法,该方法整合 了ATAC峰潜在的DNA序列信息。SANGO包括从DNA序列信息中提取细胞表示和细胞类型预测两个阶段。首先,对于所有Peak, SANGO使用通道注意力卷积神经网络从ATAC峰下的DNA序列信息中学习Peak的低维表示(见图1,顶部)。然后,SANGO通过一个全连接网络迭代地使用低维表示来重构细胞的峰统计信息。由于全连接网络学习的权重反映了表示细胞的调控模式,该权重可以被视为细胞表示。这些细胞表示将被输入到GraphTransformer模块中,通过传播共享的信息(例如细胞类型特定的峰表达模式)来消除批次效应,并学习包含在参考数据中的注释细胞标签(见图1,底部)。GraphTransformer的输出然后用于预测查询数据的细胞类型。
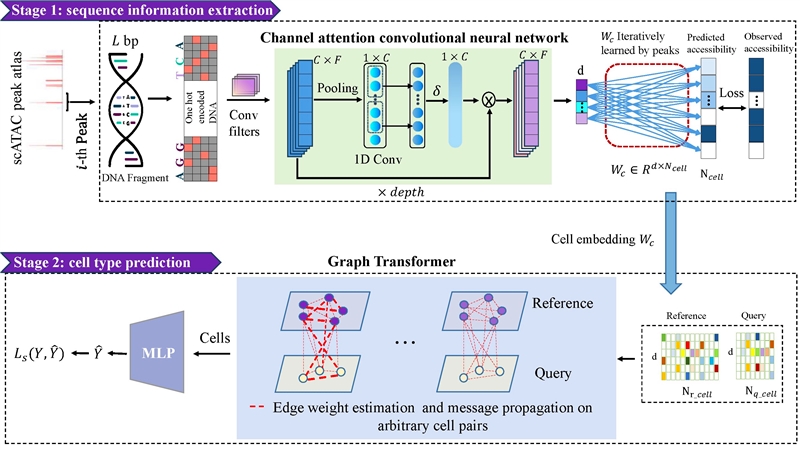
图1 SAGNO工作流程图
SANGO可有效实现跨平台和跨组织数据集的精准注释:研究人员首先在数据集内部,跨平台和跨组织数据集上验证了SANGO的性能,与传统的单细胞注释算法如Seurat和最近开发的EpiAnno和Cellcano比较,结果表明SANGO的细胞类型注释效果能展示更为精准的结果。就细胞平均分类精确度而言,SANGO相比对比方法,在跨组织上最高提高了26.3%。该结果显示SANGO能有效提取scATAC-seq数据的低维表征,并去除数据间的批次效应,实现鲁邦的分类结果。
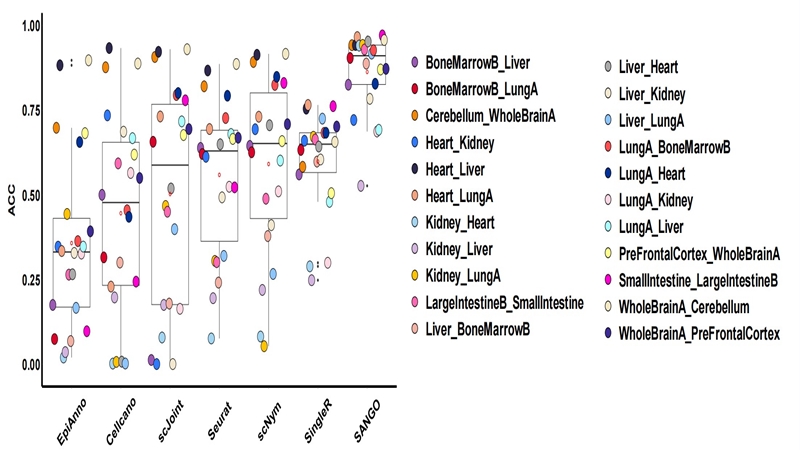
图2 在跨组织数据集上的性能
SANGO应用到大脑组织:研究人员将SANGO应用到一个大脑组织数据集上,并对数据集进行了细胞类型注释。基于注释的细胞类型,SANGO别了细胞特异的peak。基于特异Peak,研究人员进行了motif富集分析,发现每个细胞类型显示了特异的motif(图3左边)。组织特异性表达富集分析显示,与背景峰相比,在与大脑相关的组织中,SANGO识别的峰中存在着显著的细胞类型特异性基因表达富集(图3右边)。
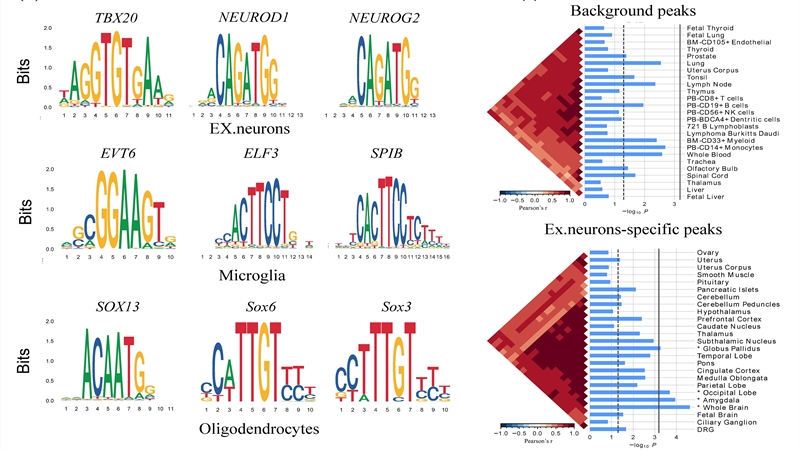
图3 SANGO促进细胞类型特异性峰值的发现,有助于转录因子结合基序富集分析和组织特异性表达富集分析
SANGO应用到癌症组织:研究人员将SANGO应用到了基底细胞癌数据中,并通过一个健康人类的图集数据集对该癌症数据集进行了注释。图4a展示的是该数据的可视化图,其中不同颜色通过真实的细胞标签着色。SANGO通过边注意力机制分数有效识别了癌症数据集中的肿瘤细胞(未知类型)。图4b中的细胞颜色代表细胞该细胞是未知细胞类型的概率,颜色越红,是未知类型的概率越高。
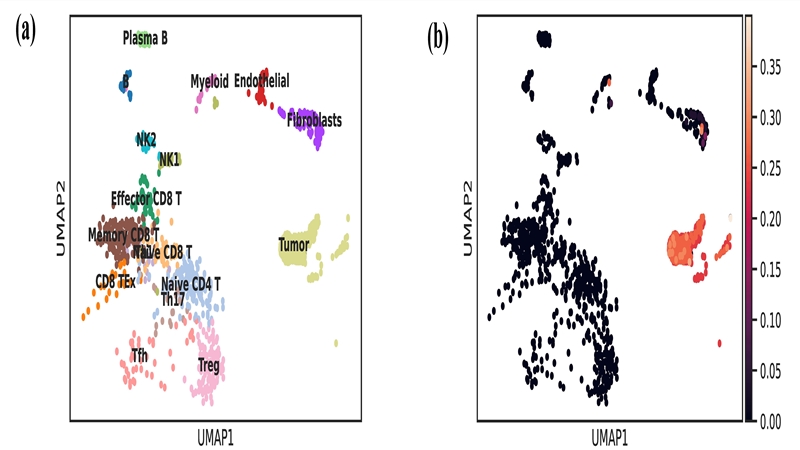
图4 SANGO识别癌症组织中的肿瘤细胞
文章链接:https://www.nature.com/articles/s43588-024-00622-7
曾远松博士毕业于中山大学,并于2023年加入重庆大学大数据与软件学院,担任弘深青年教师。他的研究方向着重于利用人工智能技术对单细胞和空间转录组数据进行分析。作为第一作者,他在多个国际期刊和会议上发表了重要论文,包括Nature Computational Science (NCS)、Bioinformatics、Briefings in Bioinformatics (BIB)、IEEE International Conference on Bioinformatics and Biomedicine (BIBM)等。
目前,曾远松博士正在大力招募对他的研究方向感兴趣的学生进行科研合作。如果你对他的研究方向感兴趣,欢迎联系邮箱:zengys@cqu.edu.cn。